효소화학 반응을 이용한 공간생물학(Spatial Biology) 연구
- 2023년 5월 4일
- 12분 분량
강명균, 이현우* | 서울대학교 화학부, rheehw@snu.ac.kr
서 론
사람의 몸은 하나의 세포에서부터 출발한다. 이 하나의 세포가 대략 60조의 딸세포로 분열하며 몸을 만드는 수많은 중간 단계에서 딸세포들은 정해진 “위치”에서 또 다른 딸세포를 만들며 장기 그리고 몸을 만든다. 또한 각 장기나 조직 안에서 여러 세포들은 “접촉”을 통한 세포간 교신을 하며, 그리고 각 세포안의 10,000 종류가 넘는 단백질들이 도메인 구조에 따라 분류되어 알맞은 세포내 “위치”로 이동되는 과정이 끊임없이 일어남으로써 우리의 삶을 이룬다. 생명현상의 각 layer에서 관찰할 수 있는 이러한 기막힌 공간 특이적인 생체 정보들은 하나의 세포 안에 모두 코딩되어 있는 것으로 생각할 수 있으며 이러한 생명체의 공간 정보를 탐색하는 새로운 학문을 “공간생물학(Spatial Biology)”라고 정의할 수 있다. 공간생물학을 연구할 수 있는 다양한 방법 중에 본 연구실에서 주도적으로 개발에 참여하고 있는 근접분자 표지기술(proximity labeling, PL)은 핵산 및 단백질 시퀀싱 방법과 접목될 수 있기 때문에 매우 풍부한 공간생물학 정보를 몇 번의 실험만으로도 획득할 수 있는 큰 혁신성이 있다. 이번 화학세계의 하이라이트 코너에서는 공간생물학 연구를 위한 근접분자 표지화학의 연구 현황 및 앞으로의 발전 방향에 관하여 논의하였다.
본 론
1. 초고해상도 근접분자 표지기술(SR-PL) 개발
생체 공간정보에 대한 연구는 단백질 수준에서 세포생물 학자 및 구조생물학자들에 의해 가장 많이 연구되어왔다. 단백질은 체내에서 세포를 구성하는 생체고분자 중 하나로 특정한 위치에서 에너지 생성, 대사물질 운반, 신호전달, 그리고 면역반응 등 중요한 역할을 수행하며 단백질-단백질 상호작용에 의해 다양한 생물학적 반응들이 일어나거나 조절된다. 단백질 상호작용 네트워크는 세포의 기능 및 생명유지에 필수적이며 이러한 상호작용이 잘못 조절될 경우 암, 신경질환을 포함한 여러 질병들을 일으키거나 세포의 노화를 촉진시킬 수 있다. 때문에 단백질간의 상호작용을 규명하는 것은 새로운 기전을 밝혀낼 뿐만 아니라 치료제를 개발하는데 새로운 지표를 제공할 수 있다.[참고문헌 1]
전통적으로 단백질 간 상호작용 규명 및 구획화된 단백질체를 알아내기 위한 방법으로는 상호 면역침전(co-im- munoprecipitation, Co-IP)방법이 많이 사용되어왔다.[참고문헌 2] 상호 면역침전 기술은 세포를 용해(lysis) 시킨 후 항체가 붙은 비드(bead)와 섞어 항원-항체 반응을 통해 특정 단백질(항원)을 분리할 수 있으며, 낮은 농도의 세정 용액을 사용하여 항원과 상호작용 단백질의 결합을 파괴하지 않고 동시에 분리해낼 수 있다. 분리한 상호작용 단백질은 트립신 분해 후 질량분석기로 분석하여 알아낼 수 있지만 이 기술은 세포 용해 이후의 단백질 상호작용에 의존하기 때문에 세포 소기관 막 등의 경계가 사라져 인위적인 결합이 일어 날 수 있다. 반대로 일시적인 결합이나 약한 결합을 이루는 단백질의 경우에는 분리 전 세정 용액에 의해 제거된다. 또한, 불용성 항원이거나 항원에 특이적인 항체가 없는 경우 사용할 수 없다는 단점들이 존재한다.[참고문헌 3] 이러한 한계점들을 보완하기 위해 약 10여 년 전에 근접분자 표지기술(proximity labeling, PL)이 개발되었으며 이 기술은 마치 효소가 스프레이 물질을 뿌리듯이 주변 단백질에 무작위로 비오틴 파생 물질을 공유결합으로 붙이는 스프레이타입 단백질 변형 효소화학 반응을 이용한다. 지금까지 PL 기술을 위하여 다양한 효소들이 개발되어왔으나 생물학자들이 선호하는 두 가지 효소가 주로 사용되고 있으며 과산화효소 (peroxidase) 기반의 APEX[참고문헌 4]와 비오틴 라이게이즈(biotin ligase) 기반의 BioID[참고문헌 5]이다. APEX는 식물에서 과산화수소를 환원시켜 항산화작용을 하는 APX라는 과산화효소의 유전자를 변형하여 활성도를 증가시켜 개발하였으며,[참고문헌 6] 약 28 kDa의 분자량으로 특정 단백질의 세포 내 위치 연구에 많이 사용하는 녹색형광단백질과 비슷한 사이즈를 가지고 있다. APEX를 이용하면 과산화수소를 통해 비오틴 페놀 (biotin-phenol, BP)을 라디칼화하여 1분이라는 빠른 시간 내에 주변의 단백질들의 표면에 있는 타이로신 잔기에 표지할(또는 비오틴화할) 수 있다[그림 1A]. BioID의 경우 대장균 안에 존재하는 비오틴 라이게이즈(BirA)의 유전자를 변형하여 만들어졌으며[참고문헌 5] 35 kDa의 분자량으로 APEX 보다 약간 큰 분자량을 갖는다. BioID는 비오틴과 아데노신 삼인산(ATP)를 이용하여 비오틴-아데노신 일인산에스터를 생성하는데 이 생성물의 에스터는 일차 아민의 친핵성 공격에 의해 아미드 결합을 형성하기 때문에 주변 단백질의 라이신을 비오틴화시킬 수 있다[그림 1A]. 이렇게 표지된 단백질들은 세포 용해 후 비오틴과 결합력이 강한 스트렙타비딘(streptavidin) 비드를 사용하여 면역침전으로 분리할 수 있으며 질량분석을 통해 동정할 수 있다.
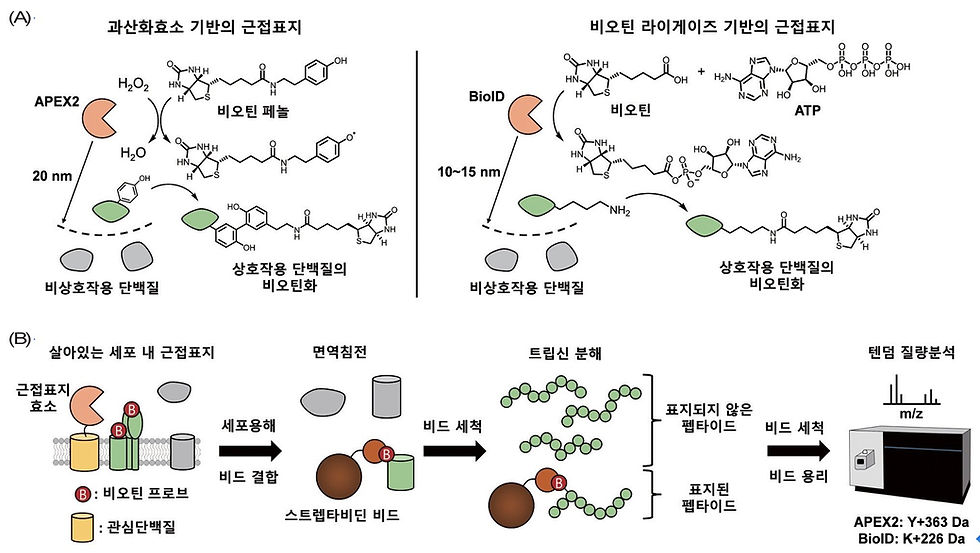
그림 1. (A) APEX와 BioID를 이용한 근접분자 표지화학 반응의 모식도와 (B) Spot-BioID를 이용한 근접분자 표지 펩타이드의 질량 분석의 모식도
APEX나 BioID를 이용한 근접분자 표지기술(PL)이 전통적인 상호 면역침전법(Co-IP) 과 가장 큰 차이를 보이는 점은 살아있는 세포에서 스프레이 반응을 진행하기 때문에 세포 용해 후에도 세포가 살아있을 때의 생리적인 공간 정보를 남길 수 있다는 점이다. 근접분자 표지기술은 처음 보고된 이후 지난 10년 동안 여러 방면에서 기술의 진보를 거듭하여 왔으며 현재는 살아있는 세포와 동물 조직에서의 공간 단백질체 정보(spatial proteomics)를 알아내는데 중요한 연구기법으로 자리잡았다. 특별히 본 연구실에서는 단백질 내 표지된 위치를 분석함으로써 근접분자 표지기술의 분석의 정확성과 공간분해능을 획기적으로 높인 초해상도 근접분자 표지기술(super-resolution proximity labeling, SR-PL)의 개발과 이를 이용한 사례에 대해 소개하고자 한다.
현재까지 많은 연구자들이 APEX 또는 BioID를 이용하여 표지된 공간단백질체 연구를 진행하여 왔으나, 대부분의 경우에는 APEX나 BioID로 비오틴화된 펩타이드만을 분석하는 것이 아닌 스트렙타비딘 비드로 분리된 단백질 전체를 분석하는 것에 초점을 맞추고 있다. 하지만 이 경우에는 비오틴화되지 않은 단백질도 다양한 이유에 의해서 마치 비오틴화 된 것처럼 여길 수 있는 오류가 있다. 이는 스트렙타비딘 비드로 면역침전 시 분자간 인력 때문에 비오틴화 되지 않은 단백질들이 비오틴화된 단백질에 붙어서 같이 분리될 수 있기 때문이다. 특별히 미토콘드리아의 경우에는 내막과 외막 단백질들이 긴밀히 연결되어 있기 때문에 미토콘드리아 기질(matrix)에서 비오틴화된 단백질을 스트렙타비딘 비드로 분리하였을 때 외막 단백질까지 같이 분리되는 경우가 빈번하게 일어난다. 또한, 세포 용해 액에서만 일어나는 매우 인공적인 단백질간 결합에 의해서 전혀 생뚱맞은 단백질이 비오틴화된 단백질에 붙어서 스트렙타비딘 비드에 살아남아서 결국 APEX나 BioID로 표지된 공간단백질체로 인식되는 경우도 있다. 이러한 경우들을 모두 고려하면 스트렙타비딘 비드로 분리된 단백질들은 절대 비오틴화된 단백질체와 같다고 여길 수 없지만 대개의 연구자들은 분석의 편의성으로 인해 스트렙타비딘 비드로 분리된 단백질을 비오틴화 단백질체로 여기는 분석방법을 따르며 이로 인해 매우 높은 레벨의 위양성(false positive) 을 포함한 낮은 해상도(low resolution)의 근접분자 표지 단백질체 정보가 얻어지게 된다.
이러한 한계를 극복하기 위해 본 연구실에서는 근접분자 표지된 펩타이드만을 정확하게 분석할 수 있는 SR-PL을 BioID와 APEX에 적용하여 개발해왔다.[참고문헌 7-11] 먼저, BioID에 의해 비오틴화된 단백질을 스트렙타비딘 비드와 결합시킨 뒤 트립신 분해를 통해서 표지되지 않은 펩타이드를 제거 하고 비오틴화 펩타이드만 비드에 남긴 뒤 열을 가해 용리 (elution)하여 분석하는 첫번째 SR-PL 기술인 Spot-BioID 방법을 개발하였다[그림 1B]. 이를 이용하여 mammalian target of rapamycin (mTOR) 단백질에서 라파마이신 (rapamycin)과 결합하는 도메인(FRB)을 따로 BioID에 붙여 인간세포에 발현시킨 뒤 상호작용 단백질체를 조사하였다.[참고문헌 7] 탠덤(tandem) 질량분석을 통해 비오틴화된 라이신 (K+226 Da)을 조사하였고, FKBP25 단백질의 80, 86, 그리고 89번째의 라이신이 라파마이신을 처리했을 때만 비오틴화가 된 것이 관측되었다. 이 결과를 기반으로 울산과학기술원 이창욱 교수연구실과 공동연구를 진행해 FKBP25-라파마이신-FRB 복합체의 결정 구조를 얻어[그림 2A] 실제로 결합하는 것을 검증하였다.[참고문헌 7]
이후 Spot-BioID 기술을 이용한 추가적인 연구에서는 미토콘드리아 외막에 존재하는 단백질인 TOMM20의 C-말단에 BioID를 붙여 상호작용 단백질을 분석하였고,[참고문헌 8] exonuclease 3′-5′domain containing 2 (EXD2) 단백질의 46, 221번째의 라이신이 비오틴화 된 것이 확인되었다[그림 2B]. 즉, EXD2 단백질이 미토콘드리아 외막에 존재하는 것을 의미하나 기존의 연구에서는 EXD2가 세포핵 또는 미토콘드리아 기질에 존재한다고 보고되어 있었다. EXD2 단백질의 세포 내 위치를 확인하기 위해 APEX의 활성도를 높인 APEX2를 이용한 전자현미경 이미징 방법을 사용하였으며, 이를 통해 N-말단 부분의 소수성인 1- 37 아미노산 서열이 EXD2가 미토콘드리아 외막에 위치하기 위한 신호서열로 작동하는 것을 밝혀내었다.[참고문헌 8] TOMM20 C-말단은 세포질로 향해 있기 때문에 EXD2의 46, 221번 째 라이신도 세포질에 위치하는 것을 파악할 수 있다. 결론적으로 질량분석 및 전자현미경 이미징 결과를 통해 EXD2 의 막 방향성(membrane topology)가 검증되었으며, 외부가수분해 효소(exonuclease)가 미토콘드리아 외막에서 세포질에 존재하는 DNA 또는 RNA 단일 가닥을 자를 것 으로 예상된다. 이에 따라 EXD2는 세포질 핵산 분해와 관련된 미토콘드리아의 알려져 있지 않은 기능과 관련되어 있을 것으로 추측하고 있다.
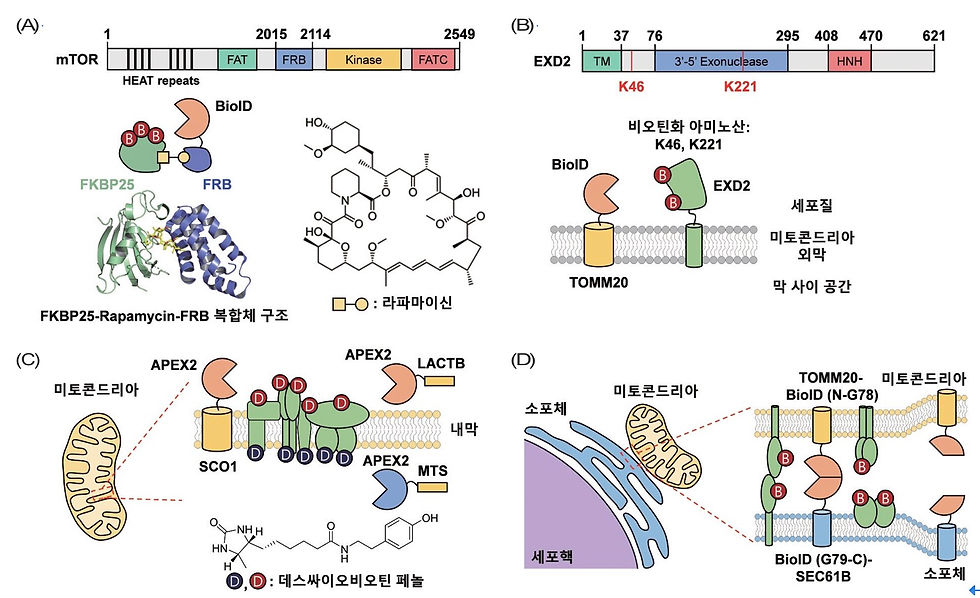
그림 2. 초고해상도 근접분자 표지기술(SR-PL)을 적용한 공간단백질체 연구의 모식도. (A) FRB의 라파마이신 의존 상호작용 단백질 분석,[참고문헌 7] (B) 미토 콘드리아외막 단백질체 연구,[참고문헌 8] (C) 미토콘드리아내막 단백질의 막 방향성 조사,[참고문헌 9] (D) 미토콘드리아와소포체의 접촉 부위에 위치하는 단백질체 연구.[참고문헌 10]
위와 같이 SR-PL 방법은 막 단백질체에 적용될 경우 근접분자 표지효소에 의해 표지된 위치에 따라 단백질의 막 방향성을 결정하는데 유용하게 사용될 수 있어 본 연구실에서는 이 방법을 미토콘드리아 내막 단백질체에 적용하였다. 미토콘드리아 내막은 높은 밀도의 단백질들로 이루어져 있으며 산화적 인산화, 대사물질 합성 등 세포 내에서 중요한 생화학적 반응이 일어나는 곳이다. 이들의 막 방향을 조사하기 위하여 APEX2를 미토콘드리아의 기질(matrix)와 이중막사이 공간 (IMS)에 각각 발현시켜[그림 2C], 미토콘드리아 내부 각 공간 단백질체를 표지하였다. 이때 APEX 에 맞는 SR-PL 방법의 개발을 위해서 새로운 프로브인 데스싸이오비오틴 페놀(desthiobiotin-phenol, DBP)을 개발하였다.[참고문헌 9] BP 경우 비오틴의 싸이오이써(thioether) 구조가 쉽게 산화가 되므로 질량분석에서 표지된 펩타이드의 신호가 약하게 나오는 반면, 황이 없는 구조의 DBP는 비오틴 산화로 인한 질량분석 신호가 감소되는 이슈를 피할 수 있다. 또한, 스트렙타비딘에 대한 데스싸이오비오틴의 결합력이 비오틴보다 상대적으로 약하기 때문에 면역침전시 용리가 잘되어 표지된 펩타이드의 수율을 높일 수 있는 장점이 있다. APEX에 의해 발생한 DBP 라디칼은 미토콘드리아 내막을 통과할 수 없기 때문에 미토콘드리아 내막에 의해 분리된 기질과 막사이공간(IMS)에 분포하는 단백질들의 타이로신 잔기들을 특이적으로 표지할 수 있음을 확인하였으며, 두 APEX construct를 통해서 총 686개의 DBP로 표지된 타이로신(Y+331 Da)의 정보를 획득할 수 있었다. 이렇게 표지된 타이로신 잔기를 갖는 펩타이드 정보들은 약 130개의 미토콘드리아 내막 단백질과 매치가 되었으며, 이 단백질들의 막 방향을 재검증 또는 새롭게 결정 및 제안하는데 사용될 수 있었다.[참고문헌 9] 또한, 본 연구실에서는 SR-PL 기술을 이용하여 미토콘드리아와 소포체의 접촉 면에 특이적으로 위치하는 단백질체의 정보도 보고하였는데, 이를 표지하기 위해 BioID 효소를 두 분자로 나누어 서로 결합할 때만 작동하는 스플릿 BioID 시스템(Contact-ID로 명명)을 개발하였다.[참고문헌 10] 4개의 조합 후보 중 N-G78/G79- C 스플릿 페어가 가장 높은 활성도를 보여 N-G78 BioID조각은 SEC61B라는 소포체 막의 단백질의 N-말단에 연결하여 세포질로 향하게 하였고, G79-C BioID 조각은 TOMM20의 C-말단에 연결하여 미토콘드리아 외막에서 세포질로 향하게 했다. 두 단백질을 동시에 발현시킨 뒤 비오틴을 16시간 처리하여 그 동안 미토콘드리아와 소포체가 인접하는 공간안의 단백질들만 표지할 수 있었으며[그림 2D], 질량분석 후 표지된 라이신을 조사하여 두 소기관이 접촉했을 때 기능을 하는 115개의 단백질을 밝혀낼 수 있었다.[참고문헌 10] 또한, 비오틴 표지된 Lysine 잔기(K+226 Da) 정보를 기반으로 115개의 단백질 중 85개의 막 단백질(73.9%, 85/115)의 막 방향성도 보고하였다. 이 중에 미토콘드리아-소포체 접촉 단백질로 새롭게 규명된 FKBP8이라는 단백질을 과발현시키면 미토콘드리아와 소포체의 접촉이 증가하는 것을 관측하였고 반대로 감소시키면 접촉도 줄어드는 것이 확인되어 FKBP8이 두 소기관의 접촉을 제어하는 중요한 역할을 한다는 것을 보고하였고 이러한 정보들은 현재 세포소기관 막 접촉 생화학을 연구하는 연구자들에게 중요한 리소스로 활용되고 있다.
2. 동물모델에서의 근접분자 표지기술 활용
근접분자 표지기술을 이용한 많은 연구들이 배양세포에서 실험을 진행하였으나 동물모델의 조직은 다양한 세포로 이루어져 있고 다세포 생물체에서 발생하는 복잡한 상호작용과 반응을 연구할 수 있어 생리학적으로 더 유의미하다. 특히 동물모델에서는 세포 간 상호작용 및 신호전달, 세포 외 기질의 영향과 같은 세포밖의 환경을 제공할 수 있으며, 질병 진행 및 약물 효능과 같은 복잡한 생물학적 과정을 연구할 때 배양세포에서는 확인할 수 없는 중요한 생물학 정보를 알아낼 수 있다. 이러한 차이점에 의해 실제로 미토콘드리아와 같은 세포소기관 단백질체에 관한 실험도 배양세포와 동물모델의 조직에서 실험할 때 사뭇 다른 결과를 도출할 수 있다. 현재 Human protein atlas,12 G-TEx Por- tal13과 같은 동물모델에서 장기 별 유전자의 발현 정보를 포함한 데이터베이스들이 구축 되어있지만 이 정보는 유전자의 발현양(expression level) 및 결실 표현형(deficient phenotype)에 관한 정보들이 주를 이루고 있을 뿐, 각 유전자가 각 조직에서 발현된 단백질의 위치 및 상호작용 단백질 등의 공간생물학에 해당하는 정보는 여전히 부족하다고 볼 수 있다.

그림3.SEC61B-BioID 쥐와 MTS-APEX 쥐를 각각 이용한 (A) 조직 특이적인 간(liver) 분비단백질체 연구[참고문헌 16]와 (B) 근육 특이적인 미토콘드리아 기질 단백질체 연구[참고문헌 17]의 모식도
이러한 필요성에 의해 연구자들은 초파리,[참고문헌 14] 예쁜꼬마선충,[참고문헌 15] 쥐[참고문헌 16,17]에서 근접분자 표지기술법을 적용하여 기존의 방법으로 알아내기 힘들었던 조직특이적인 공간단백질체에 관한 정보를 알아내고 있다. 특별히 조직에서 분비되는 분비단백질은 세포 안에서 생성된 후 외부로 분비되어 혈관을 통해 다양한 생화학 반응을 수행하며 장기간 커뮤니케이션에 핵심적인 역할을 한다. 본 연구실에서는 조직특이적인 분비 단백체를 밝혀내기 위해 또 다른 근접분자 표지효소인 TurboID를 SEC61B 단백질의 C-말단에 부착시켜 소포체 내강으로 향하게 하였다.[참고문헌 16] 이 연구에서는 당시에 개발된 TurboID를 사용하였는데, BioID에 유전자 변형을 시켜 활성을 극대화하여 개량된 효소이다.[참고문헌 18] 이 단백질을 쥐의 간에 발현시키기 위해서 쥐의 꼬리혈관에 정맥 주사를 통해 SEC61B-TurboID 유전자를 삽입한 아데노 부속 바이러스를 간으로 전달하여 SEC61B-TurboID를 발현시켰으며 3일 동안 24 mg/kg의 양으로 비오틴을 공급해 표지를 진행하였다[그림 3A]. 웨스턴 블랏 실험을 통하여 쥐의 간에 SEC61B-TurboID를 발현시키고 biotin 을 공급하였을 때에만 쥐의 혈장에 비오틴 표지된 단백질을 관찰할 수 있었고, SR-PL 기술을 적용하여 약 50개의 간 조직의 분비단백질이 비오틴 표지된 것을 확인하였다.[참고문헌 16] 또한 인슐린 저항성을 유발한 쥐에도 같은 실험을 진행하여 당뇨병 상황에서 간 분비 단백질이 매우 상이하게 달라짐을 역시 SR-PL 방법을 적용하여 알아낼 수 있었다.
SR-PL 방법은 쥐의 조직별 미토콘드리아 기질 단백질체를 알아내는데에도 활용되었다. 지금까지는 이를 알아내기 위해 수크로스를 이용한 기울기 원심분리법을 적용하여 미토콘드리아를 분리한 뒤 약한 세정제를 이용해 외막을 파괴하는 방법이 사용되어져 왔다. 하지만 이 방법은 많은 불순물을 포함하며 미토콘드리아 단백질이 아닌 다른 세포 소기관 단백질도 혼합되는 큰 한계점이 존재한다. 이에 본 연구실에서는 쥐의 조직에 미토콘드리아 기질 특이적으로 APEX2를 발현시킬수 있는 MTS-APEX2 유전자를 이식 한 쥐(MAX-Tg 쥐)를 제작하여 조직특이적인 미토콘드리아 기질 단백질체 맵핑 프로젝트를 시작하였다. MAX-Tg 쥐에서 심장, 전경골근, 비장근을 추출하여 SR-PL 분석을 위해 DBP와 과산화수소를 이용하여 표지한 후 분석하 였으며[그림 3B] 각 근육 장기에서 193, 237, 그리고 246 개의 DBP로 표지된 단백질을 발견하였다.[참고문헌 17] 이 단백질들은 대부분 미토콘드리아 기질 단백질(88.0% 이상)이었으 며, 이 중에 RTN4IP1 이라 불리는 단백질은 이전 연구에 서는 미토콘드리아 외막 단백질로 보고되었으나 본 연구에 서는 전자현미경 기법을 이용하여 RTN4IP1이 미토콘드리아 기질에 있는 것을 증명하였다. RTN4IP1의 구조 연구를통해 이 단백질이 미토콘드리아 기질에서 NADPH와 결합 하여 퀴논 등의 전자수용체로의 전자 전달 및 Coenzyme Q 생합성에 중요한 역할을 담당하는 단백질임을 알아낼 수 있었다.
3. 광학가교반응을 이용한 근접분자 가교기술(proximity crosslinking)
근접분자 표지기술은 현재 공간단백질체 연구(Spatial Proteomics)에 활용되는 중요한 연구기법으로 자리잡았지만, APEX나 BioID가 만들어내는 비오틴 스프레이 물질의 표지 반경이 수십 나노미터에 이르기 때문에 세포소기관 수준의 단백질체를 분석하는데 있어 최적화되어 있지만 많은 분자생물학자들이 관심을 갖는 단백질-단백질간 결합(interactome)을 분석하는 데에는 다소 넓은 반응 반경을 갖고 있고 이를 정확히 분석하려면 엄격한 대조군 실험 들이 필요하다. 이에 따라 본 연구실에서는 APEX, BioID 와 같은 스프레이 화학반응이 아닌 광학가교결합 기반의 새로운 근접분자 가교기술(proximity crosslinking)을 개발하였다.[참고문헌 19] 광학가교결합을 일으키는 물질을 특정 단백질에 표적화 시키기 위하여 할로택 단백질이 이용되었으며, 이와 공유결합을 이룰 수 있는 염화핵실(hexyl chloride) 구조를 포함한 광학가교물질(VL1)을 합성하였다. 광학가교물질로 사용된 4-azido-Nethyl-1,8-naphthalimide (AzNP)는 가시광선 영역대인 청색광을 이용해 나이트렌 (nitrene)을 생성할 수 있으며 기존의 UV에 의해서 활성화되는 광학가교물질(예를 들어, 아릴아자이드, 벤조페논, 다이아지린)과 비교하여 세포 독성이 적다고 볼 수 있다 [그림 4]. 나이트렌은 주변 단백질과 C-H 또는 N-H 삽입을 통한 가교결합을 이룰 수 있으며 발생된 나이트렌은 여전히 할로택 단백질에 묶여있기 때문에 해당 단백질과 접촉할 수 있는 거리의 단백질과만 반응할 수 있도록 반응 설계되어있다. 이 기술을 이용하여 SARS-CoV-2 바이러스의 유전체에 암호화 되어있는 구조 단백질인 뉴클레오캡시드(N) 과 할로택 단백질을 결합시킨 N-HaloTag 단백질을 인간세포에 발현시켰을 때 이 바이러스 단백질과 결합 하는 인간세포 유래 단백질을 따로 분리하여 질량 분석하 였다.[참고문헌 19] 질량분석 결과 22개의 결합 단백질이 밝혀졌으며,

그림 4. 광학가교물질(VL1)을 이용한 근접분자 가교기술(proximity crosslinking)[참고문헌 19]의 모식도
이중 11개의 단백질이 RNA 결합 단백질이고, 이 중에 8개의 단백질이 유전자 발현을 억제해 항바이러스 기능을 갖는다고 알려진 스트레스 과립(stress granule)의 구성 단백질임을 알게 되었다. 이 결과는 사람의 세포 안의 소기관인 스트레스 과립이 SARS-CoV-2 바이러스 면역반응에 중요한 기능을 할 수 있음을 시사한다.
결 론
앞으로의 전망
2002년 노벨생리학상 수상자인 Sydney Brenner는 “Progress in science depends on new techniques, new discoveries and new ideas, probably in that order" 라고 언급하며, 과학의 발전에 있어 새로운 연구 기술 개발의 중요성을 강조하였다. 효소화학 반응에서 출발한 PL 기술 은 현재까지 주로 단백질 및 핵산과 같은 생체고분자를 표적화 하여 연구가 진행되었으나, PL 효소를 통해 생성된 반응성이 강한 비오틴 파생 물질은 생체고분자와만 반응하는 것이 아니라 같은 공간에 존재하는 대사물질(metabolite)과도 반응할 수 있음을 본 연구실의 최근 연구를 통하여 확인하였다. 특별히 대사물질은 대부분 단분자 물질로 이루어졌기 때문에 생체고분자를 형광 이미징하는 방법 (GFP 태깅, 항체 부착 등)을 적용하기가 어렵고 단분자를 분리하고 관찰하는 기술들은 생명과학의 주요 연구 분자인 단백질이나 핵산과 같은 생체고분자를 다루는 방법과 완전히 다르기 때문에 대사체에 관한 연구는 앞으로 생물학 연구에서 많은 발전이 이루어질 수 있는 영역이라고 볼 수 있 다[그림 5]. 이러한 상황에서 PL 방법을 이용한 공간대사체 맵핑을 할 수 있다면 이는 지금까지 알려져 있지 않은 대사물질의 공간생물학을 연구할 수 있는 혁신적인 연구방법이 될 수 있으며,[참고문헌 20] 이에 대한 연구는 현재 삼성미래기술 육성재단의 지원을 받고 수행하고 있다.
또한, PL 효소를 특정세포의 세포외공간에 유전적 방법 으로 발현시키면 특정세포와 인접한 다른 세포막 단백질을 비오틴화 시킬 수 있으며 이러한 세포만 분리하여 단일세포를 시퀀싱하면 특정세포와 인접한 세포가 무엇인지를 알아낼 수 있는 생체 내 세포 배치 지도를 그릴 수 있는 중요한 툴이 될 수 있다.[참고문헌 20] 이처럼 PL 기술은 살아있는 세포 안의 대사체의 분포와 같은 nanoscale에서 부터 동물의 조직안의 세포 네트워크와 같은 miliscale의 공간생물학 정보를 모두 알아낼 수 있는 중요한 툴이 될 것으로 예상하며 향후 10년간 PL 기술을 통해서 다양한 레벨의 풍부한 공간 생물학 정보가 축적될 것으로 내다본다[그림 5]. 또한, 이렇게 축적된 공간분자체 정보는 같은 공간 안에 존재하는 분자들간의 네트워킹을 모델링하는데 유용하게 사용될 수 있을 것이며 이러한 연구를 통해 각 생체내 공간에 위치한 분자들이 어떻게 서로 모이고 기능하는지에 대한 새로운 차원의 이해가 가능할 것으로 예상한다. PL 기술을 통하여 몸의 각 장기, 장기안의 서로 다른 세포타입, 그리고 그 서로 다른 세포타입 안의 각 세포소기관 별로 공간분자체 정보들이 축적되었을 때 우리는 하나의 세포 그리고 하나의 몸 이라는 복잡하고 정교한 시스템이 어떻게 이루어져 있고 어떻게 기능하는지를 조금 더 분명히 이해할 수 있을 것으로 예상한다.

그림 5. 근접분자 표지기술을 이용한 다양한 레벨의 공간생물(Spatial Biology) 연구[참고문헌 20]의 모식도
참고문헌
1. Lu, H. et al. “Recent advances in the development of protein-protein interactions modulators: mechanisms and clinical trials.” Signal Transduct Target Ther 2020, 5, 213, doi:10.1038/s41392-020-00315-3.
2. Phizicky, E. M. & Fields, S. “Protein-protein interactions: methods for detection and analysis.” Microbiol. Rev. 1995, 59, 94−123, doi:10.1128/mr.59.1.94-
123.1995.
3. Qin, W., Cho, K. F., Cavanagh, P. E. & Ting, A. Y. “Deciphering molecular interactions by proximity labeling.” Nat. Methods 2021, 18, 133−143, doi:10.1038/ s41592-020-01010-5.
4. Rhee, H. W. et al. “Proteomic mapping of mitochondria in living cells via spatially restricted enzymatic tagging.” Science 2013, 339, 1328-1331, doi:10.1126/
science. 1230593.
5. Roux, K. J., Kim, D. I., Raida, M. & Burke, B. “A promiscuous biotin ligase fusion protein identifies proximal and interacting proteins in mammalian cells.” J.Cell. Biol. 2012, 196, 801−810, doi:10.1083/jcb.201112098.
6. Martell, J.D. et al. “Engineered ascorbate peroxidase as a genetically encoded reporter for electron microscopy.” Nat. Biotechnol. 2012, 30, 1143−1148, doi:10.1038/nbt.2375.
7. Lee, S. Y. et al. “Proximity-Directed LabelingReveals a New Rapamycin-Induced Heterodimer of FKBP25 and FRB in Live Cells.” ACS Cent. Sci. 2016, 2, 506−516, doi:10.1021/acscentsci.6b00137.
8. Park, J. et al. “The structure of human EXD2 reveals a chimeric 3' to 5' exonu- clease domain that discriminates substrates via metal coordination.” Nucleic Acids Res. 2019, 47, 7078-7093, doi:10.1093/nar/gkz454.
9. Lee, S. Y. et al. “Architecture Mapping of the Inner Mitochondrial Membrane Proteome by Chemical Tools in Live Cells.” J. Am. Chem. Soc. 2017, 139, 3651−3662, doi:10.1021/jacs.6b10418.
10. Kwak, C. et al. “Contact-ID, a tool for profiling organelle contact sites, reveals regulatory proteins of mitochondrial-associated membrane formation.” Proc. Natl. Acad. Sci. U. S. A. 2020, 117, 12109-12120, doi:10.1073/pnas.1916584117.
11. Kang, M. G. & Rhee, H. W. “Molecular Spatiomics by Proximity Labeling.” Acc.Chem. Res. 2022, 55, 1411−1422, doi:10.1021/acs.accounts.2c00061.
12. Uhlen, M. et al. Proteomics. “Tissue-based map of the human proteome.” Sci- ence 2015, 347, 1260419, doi:10.1126/science.1260419.
13. Consortium, G. T. “The Genotype-Tissue Expression (GTEx) project.” Nat. Genet
2013, 45, 580−585, doi:10.1038/ng.2653.
14. Mannix, K. M., Starble, R. M., Kaufman, R. S. & Cooley, L. “Proximity labeling reveals novel interactomes in live Drosophila tissue.” Development 2019, 146, doi:10.1242/dev.176644.
15. Reinke, A. W., Mak, R., Troemel, E. R. & Bennett, E. J. “In vivo mapping of tissue- and subcellular-specific proteomes in Caenorhabditis elegans.” Sci. Adv.2017, 3, e1602426, doi:10.1126/sciadv.1602426.
16. Kim, K. E. et al. “Dynamic tracking and identification of tissue-specific secretory proteins in the circulation of live mice.” Nat.Commun. 2021, 12, 5204, doi:10.1038/ s41467-021-25546-y.
17. Park, I. et al., doi:10.1101/2021.10.14.464368 (2021).
18. Branon, T. C. et al. “Efficient proximity labeling in living cells and organisms with TurboID.” Nat. Biotechnol. 2018, 36, 880−887, doi:10.1038/nbt.4201.
19. Mishra, P. K. et al. “A chemical tool for blue light-inducible proximity photo-crosslinking in live cells.” Chem. Sci. 2022, 13, 955−966, doi:10.1039/d1sc04871f.
20. Choi, C. R. & Rhee, H. W. “Proximity labeling: an enzymatic tool for spatial biol- ogy.” Trends Biotechnol 2022, 40, 145−148, doi:10.1016/j.tibtech.2021.09.008.

강 명 균 Myeong-Gyun Kang
• 울산과학기술원 의생명과학, 학사 (2009-2014)
• 울산과학기술원화학과, 박사
(2014-2020, 지도교수 : 이현우, 권태혁)
• 서울대학교화학분자공학 교육연구단,
박사 후 연구원(2020-현재, 지도교수 : 이현우)

이 현 우 Hyun-Woo Rhee
• 서울대학교 생명과학부/화학부, 학사 (2000-2004, 복수전공)
• 서울대학교 화학부, 박사(2004-2009, 지도교수 : 홍종인)
• 서울대학교 화학부, 박사 후 연구원 (2009-2010, 지도교수 : 홍종인)
• MIT 화학과, 박사 후 연구원(2010-2012, 지도교수 : Alice Ting)
• 울산과학기술원화학과, 조교수/부교수(2013-2018)
• 서울대학교화학부, 부교수(2018-현재)





댓글